In diesem Blogbeitrag werden wir uns mit Histogrammen beschäftigen.
Zunächst müssen wir die nötigen Bibliotheken laden, also ggplot, und für die Beispiele den Beispieldatensatz palmerpenguins:
library(ggplot2) library(palmerpenguins)
Einfacher Histogramm-Plot der Flossenlänge
Beginnen wir mit einem einfachen Histogramm-Plot der Flossenlänge der Pinguine:
ggplot(penguins, aes(x=flipper_length_mm)) + geom_histogram()
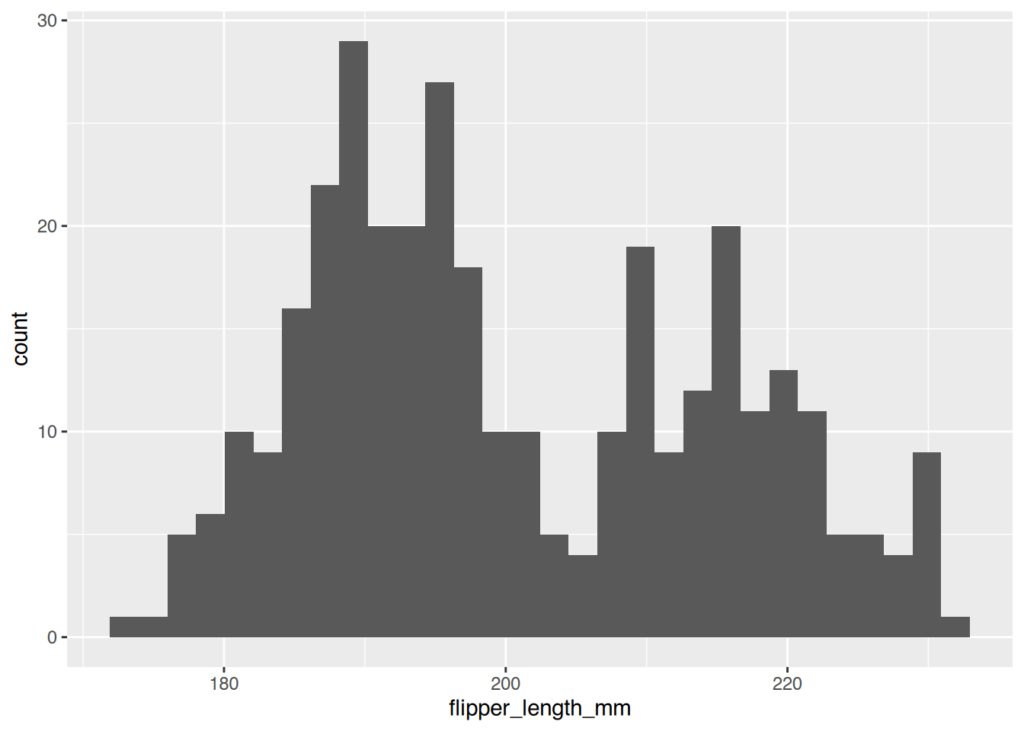
Dieser Plot zeigt die Verteilung der Flossenlängen aller Pinguine im Datensatz.
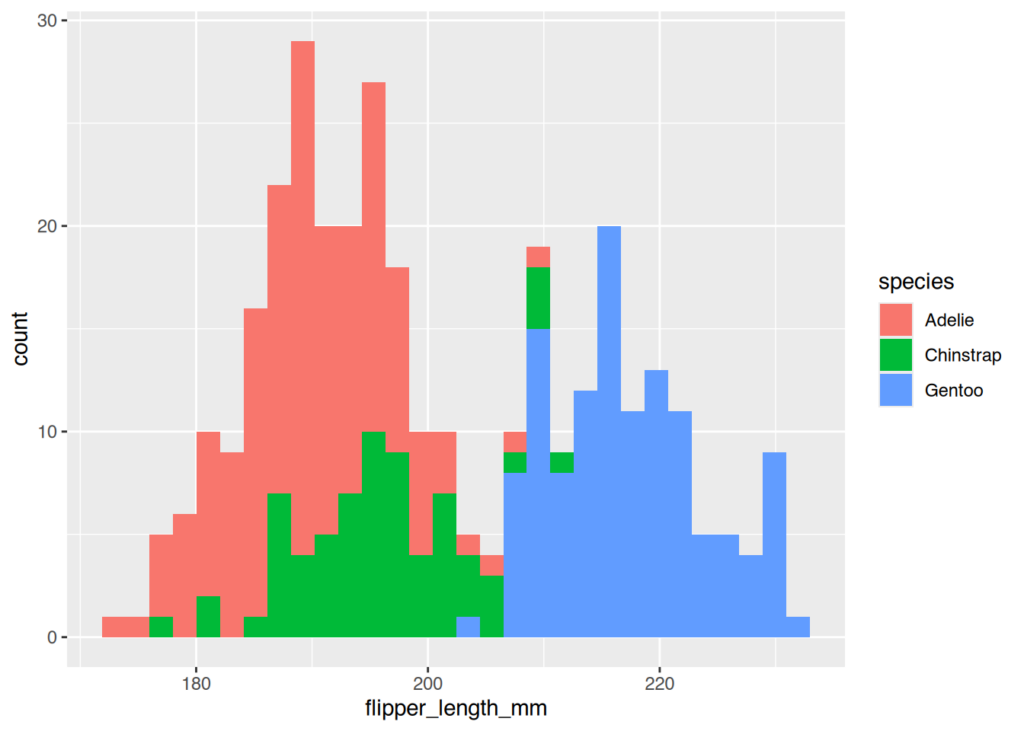
Histogramm-Plot mit farblicher Unterscheidung nach Pinguinarten
Um die Verteilung der Flossenlängen nach Pinguinarten zu unterscheiden, fügen wir die fill-Ästhetik hinzu:
ggplot(penguins, aes(x=flipper_length_mm, fill=species)) + geom_histogram()
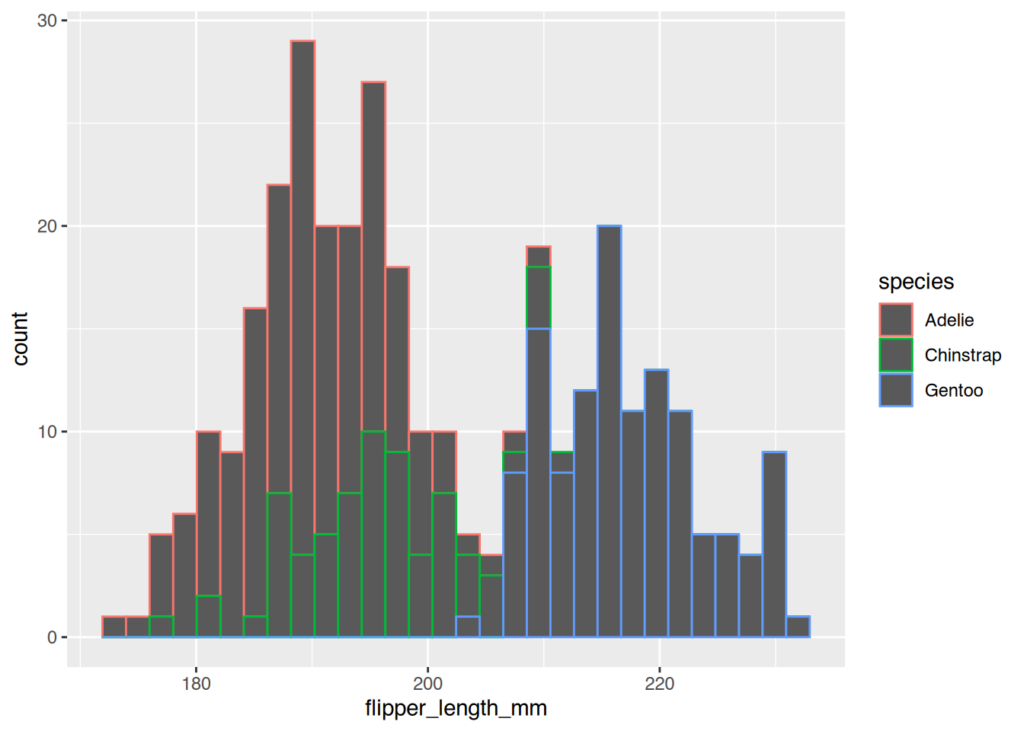
Bei Histogrammen wird zwischen der Füllfarbe (mit fill) und der Farbe der Balkenumrandung unterschieden. Würde man also color nehmen, dann würde nur die Balkenumrandung angepasst werden:
ggplot(penguins, aes(x=flipper_length_mm, color=species)) + geom_histogram()
Histogramm-Plot mit benutzerdefinierten Farben für die Pinguinarten
Um die Farben der Balken manuell festzulegen, verwenden wir scale_fill_manual:
ggplot(penguins, aes(x=flipper_length_mm, fill=species)) +
geom_histogram() +
scale_fill_manual(values = c("darkorange", "darkgreen", "purple")) +
labs(
x = "Flossenlänge [mm]",
y = "Anzahl"
)
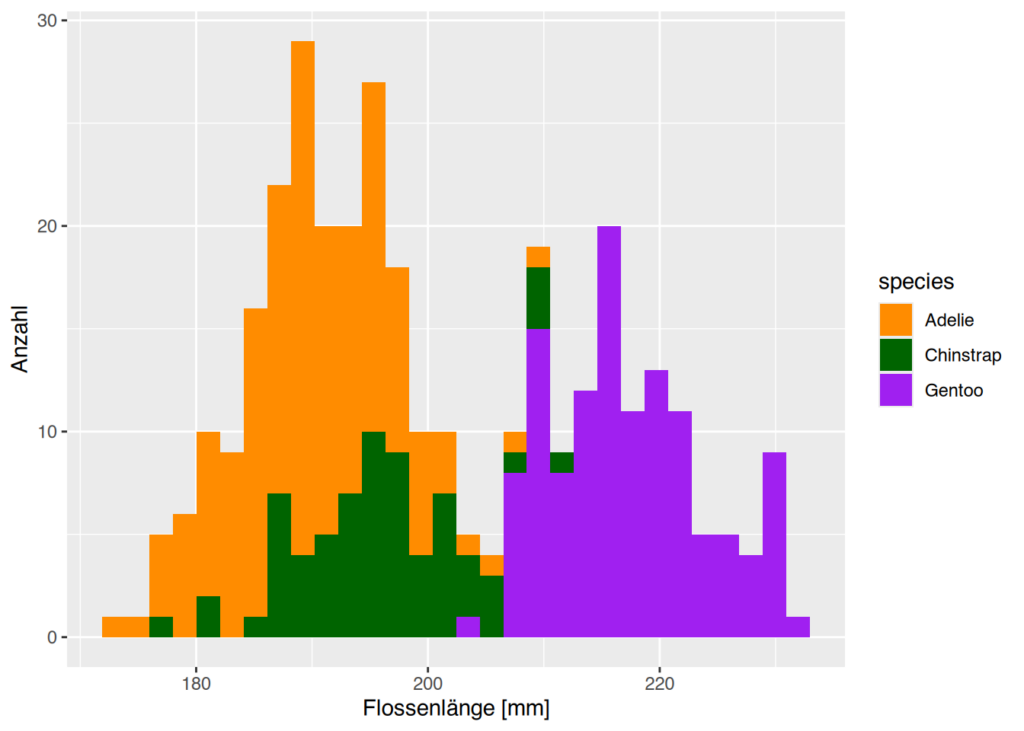
Bei diesem Beispiel sieht man, warum eine Umrandung durchaus nützlich sein kann. Wenn alle Balken dieselbe Umrandungsfarbe haben sollen, muss das direkt in geom_histogram() definiert werden.
ggplot(penguins, aes(x=flipper_length_mm, fill=species)) +
geom_histogram(color="black") +
scale_fill_manual(values = c("darkorange", "darkgreen", "purple")) +
labs(
x = "Flossenlänge [mm]",
y = "Anzahl"
)
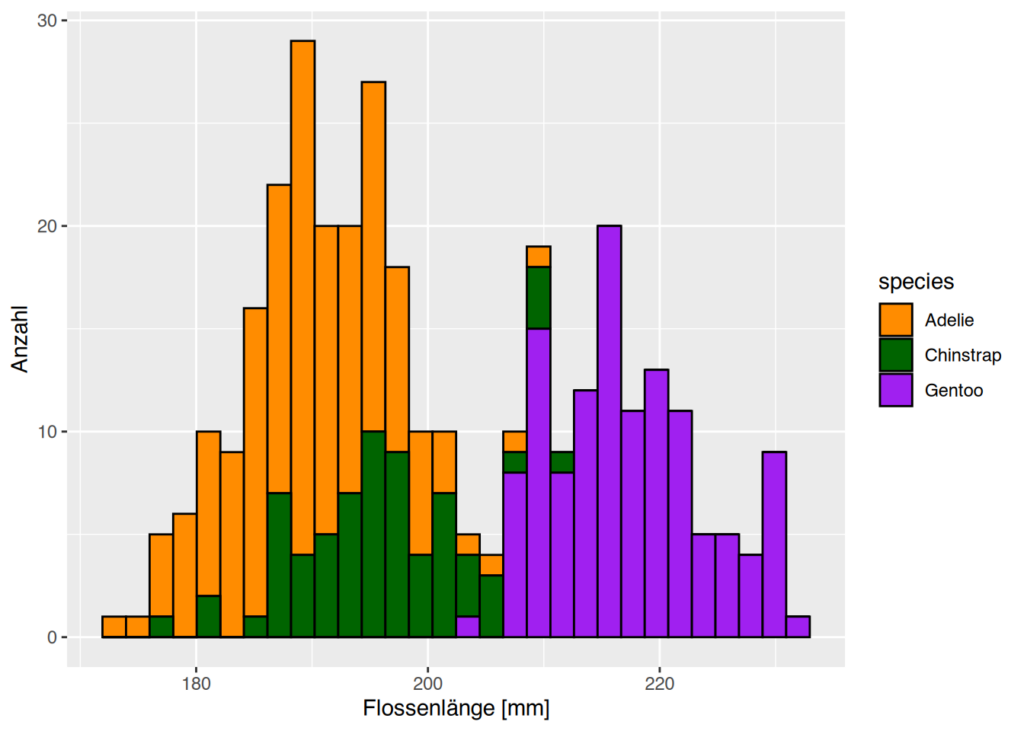
Daten von verschiedenen Pinguinarten vergleichen
Wenn man die Histogramme der Pinguinarten vergleichen möchte, dann kann man mit facet_grid() dafür sorgen, dass alle Histogramme übereinander sind, die x-Achse ist gleich für alle Plots, und auch die y-Achse ist gleich, dann sind Balken vergleichbar. species~. heißt dabei, dass die Spezies auf der y-Achse übereinander gelegt wird. Wenn man die Plots nebeneinander haben wollen würde, könnte man .~species verwenden.
ggplot(penguins, aes(x=flipper_length_mm)) +
geom_histogram(color="black") +
facet_grid(species~.)+
labs(
x = "Flossenlänge [mm]",
y = "Anzahl"
)
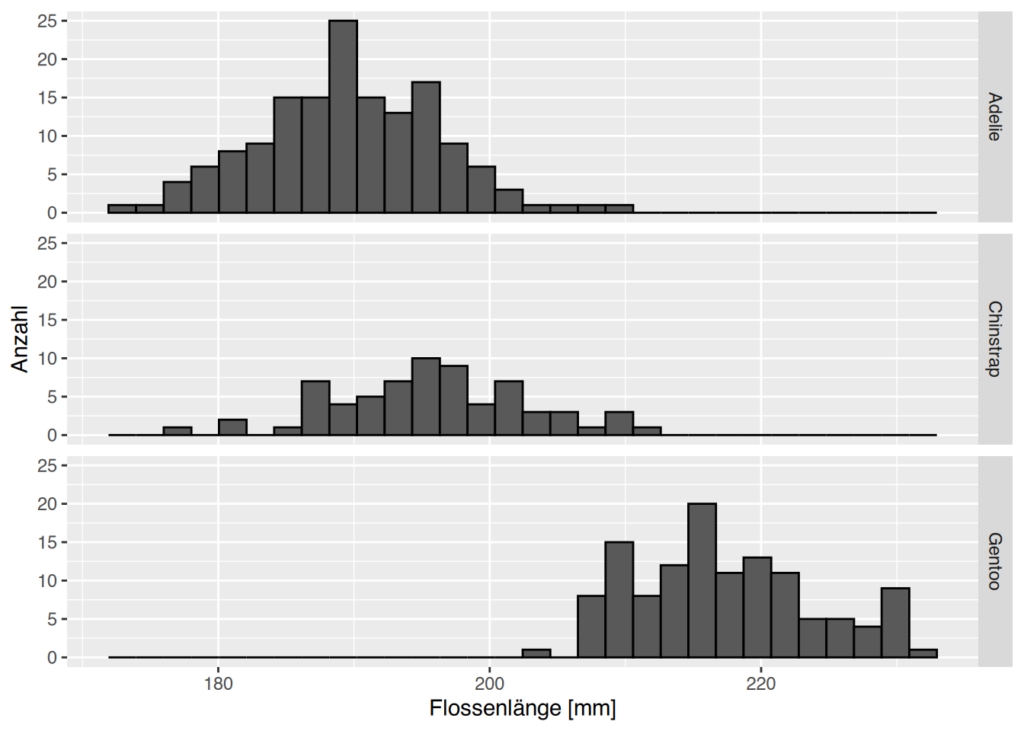
Histogramm-Plot mit Dichtekurve
Um die Dichte der Flossenlängenverteilung zu visualisieren, fügen wir eine Dichtekurve hinzu und skalieren die y-Achse auf Dichte:
ggplot(penguins, aes(x=flipper_length_mm)) +
geom_histogram(aes(y= ..density..), color="black") +
geom_density(color="darkred") +
labs(
x = "Flossenlänge [mm]",
y = "Dichte"
)
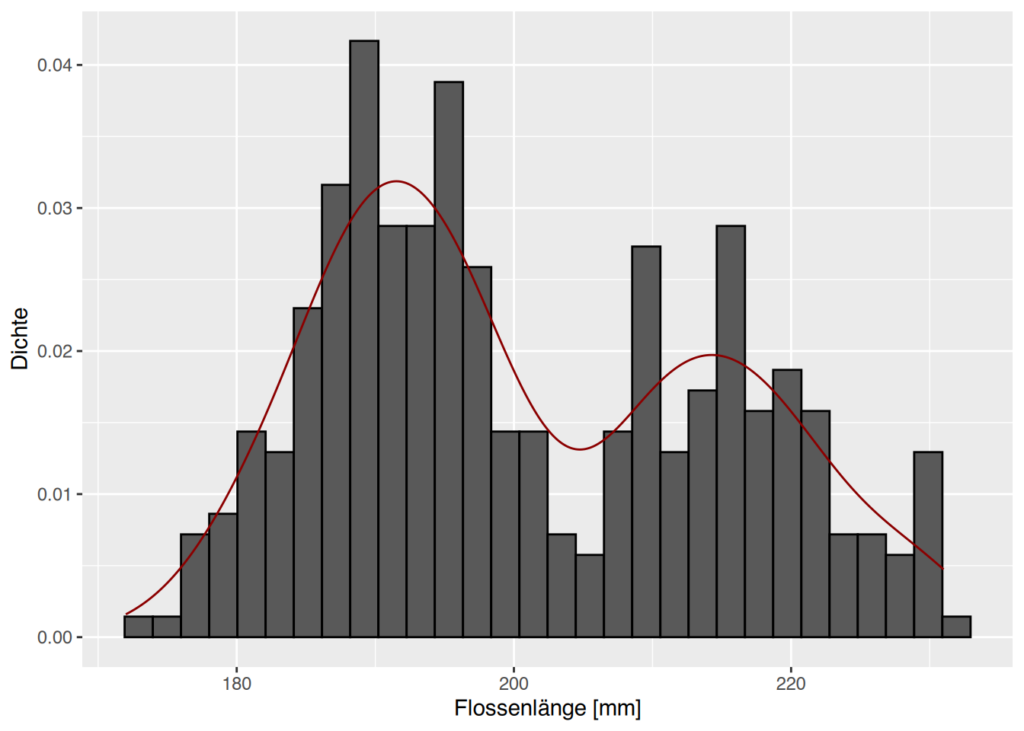
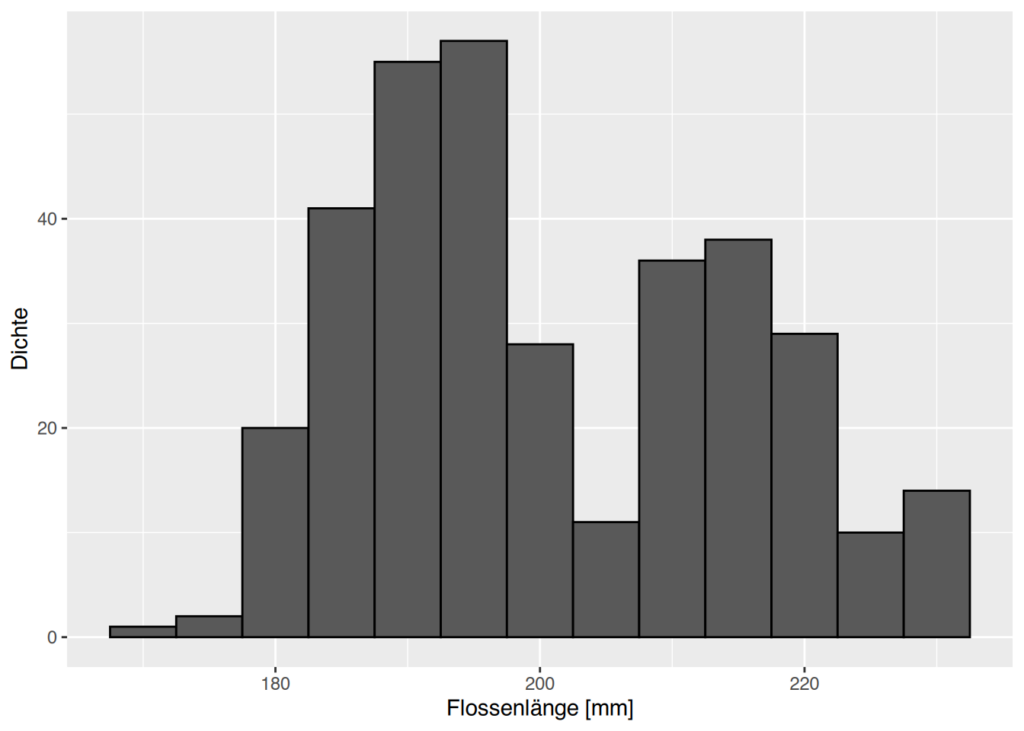
Balkenbreite anpassen
Schließlich können wir die Breite der Balken(bins) anpassen, um die Granularität des Histogramms zu ändern:
ggplot(penguins, aes(x=flipper_length_mm)) +
geom_histogram(color="black", binwidth=5) +
labs(
x = "Flossenlänge [mm]",
y = "Dichte"
)